Approssimazione di una curva parametrica nello spazio con TensorFlow
Questo post fa parte di una serie di post sull'approssimazione di oggetti matematici (funzioni, curve e superfici) tramite una rete neurale di tipo MLP (percettrone multistrato, dall'inglese Multi-Layer Perceptron);
per una introduzione sull'argomento si veda il post Approssimazione con percettroni multistrato altamente configurabili.
L'argomento di questo post è l'approssimazione di una curva parametrica, con parametro $t$ appartenente a un intervallo chiuso, su un sistema di riferimento 3-D $Oxyz$ definita con una terna di funzioni $x(t) \colon [a,b] \to \rm I\!R$, $y(t) \colon [a,b] \to \rm I\!R$ e $z(t) \colon [a,b] \to \rm I\!R$
le quali restituiscono rispettivamente il valore della coordinata $x$, della coordinata $y$ e della coordinata $z$ al variare del parametro $t$;
equivalentemente una curva parametrica nello spazio è definita anche con una funzione vettoriale $f(t) \colon [a,b] \to {\rm I\!R x \rm I\!R x \rm I\!R}$ così definita: $$f(t) = \begin{bmatrix} x(t) \\ y(t) \\ z(t) \\ \end{bmatrix}$$
ove le tre componenti dei vettori dell'immagine della funzione rappresentano rispettivamente le coordinate $x$, $y$ e $z$ della curva nello spazio.
La seconda definizione suggerisce una architettura di un MLP ove il layer di input contiene un solo neurone in quando la dimensione del dominio è 1 (l'intervallo $[a,b]$)
mentre il layer di output contiente 3 neuroni in quanto la dimensione del codominio è 3.
Nell'ambito dell'approssimazione di una curva parametrica continua e limitata nello spazio, lo scopo di questo post è di consentire all'utilizzatore di testare diverse combinazioni di architettura MLP,
funzioni di attivazione, algoritmo di addestramento e funzione di loss senza scrivere codice ma agendo solamente sulla linea di comando di quattro script Python che implementano separatamente le seguenti funzionalità:
- Creazione dei dataset
- Definizione dell'architettura del MLP + Addestramento
- Predizione
- Visualizzazione del risultato
Per ottenere il codice si veda il paragrafo Download del codice completo in fondo a questo post.
Lo stesso identico meccanismo è stato realizzato usando la tecnologia PyTorch; si veda il post Approssimazione di una curva parametrica nello spazio con PyTorch pubblicato sempre su questo sito web.
Creazione dei dataset
Scopo del programma Python pmc3t_gen.py
è di generare i dataset (di training e/o di test) da utilizzare nelle fasi successive;
prende in linea di comando le tre funzioni componenti da approssimare (in sintassi lambda body), l'intervallo del parametro indipendente $t$ (inizio, fine e passo di discretizzazione)
e genera il dataset in un file nel formato csv applicando le tre funzione all'intervallo del parametro $t$ passato.
Il file csv in uscita ha infatti quattro colonne (senza header): la prima colonna contiene i valori, ordinati in modo crescente, del parametro indipendente $t$ nell'intervallo desiderato con il passo di discretizzazione specificato;
la seconda colonna, terza e quarta colonna contengono rispettivamente i valori delle componenti $x$, $y$ e $z$, ovverosia i valori delle funzioni $x(t)$, $y(t)$ e $z(t)$ corrispondenti al valore di $t$ della prima colonna.
Per ottenere l'usage del programma è sufficiente eseguire il seguente comando:
$ python pmc3t_gen.py --helpusage: pmc3t_gen.py [-h]
-h, --help show this help message and exit
--dsout DS_OUTPUT_FILENAME dataset output file (csv format)
--xt FUNCX_T_BODY x=x(t) body (lamba format)
--yt FUNCY_T_BODY y=y(t) body (lamba format)
--zt FUNCY_T_BODY z=z(t) body (lamba format)
--rbegin RANGE_BEGIN begin range (default:-5.0)
--rend RANGE_END end range (default:+5.0)
--rstep RANGE_STEP step range (default: 0.01)Un esempio di uso del programma pmc3t_gen.py
Si supponga di voler approssimare la spirale di Archimede nell'intervallo $[0.0,20.0]$ stirata lungo $z$ descritta dalla seguente funzione $$f(t) = \begin{bmatrix} x(t)=\frac{1}{10} t \cos t \\ y(t)=\frac{1}{10} t \sin t \\ z(t)=t \\ \end{bmatrix}$$ Tenendo presente che np è l'alias della libreria NumPy, le tre espressioni componenti si traducono in sintassi lambda body Python così:
0.1 * t * np.cos(t)
0.1 * t * np.sin(t)
t$ python pmc3t_gen.py \
--dsout mytrain.csv \
--xt "0.1 * t * np.cos(t)" \
--yt "0.1 * t * np.sin(t)" \
--zt "t" \
--rbegin 0.0 \
--rend 20.0 \
--rstep 0.01$ python pmc3t_gen.py \
--dsout mytest.csv \
--xt "0.1 * t * np.cos(t)" \
--yt "0.1 * t * np.sin(t)" \
--zt "t" \
--rbegin 0.0 \
--rend 20.0 \
--rstep 0.0475Definizione dell'architettura del MLP + Addestramento
Questa funzionalità ha due implementazioni differenti secondo le due definizioni matematiche di curva parametrica: l'implementazione ufficiale è quella che approssima con un solo MLP la curva definita come funzione vettoriale.
La versione ufficiale è realizzata dal programma Python pmc3t_fit.py
che, in accordo con i parametri passati in linea di comando, crea dinamicamente un MLP ed effettua il suo addestramento al fine di approssimare la funzione vettoriale che definisce la curva sul piano.
Per ottenere l'usage della versione ufficiale del programma è sufficiente eseguire il seguente comando:
$ python pmc3t_fit.py --helpusage: pmc3t_fit.py [-h]
--trainds TRAIN_DATASET_FILENAME
--modelout MODEL_PATH
[--epochs EPOCHS]
[--batch_size BATCH_SIZE]
[--hlayers HIDDEN_LAYERS_LAYOUT [HIDDEN_LAYERS_LAYOUT ...]]
[--hactivations ACTIVATION_FUNCTIONS [ACTIVATION_FUNCTIONS ...]]
[--optimizer OPTIMIZER]
[--loss LOSS]Si rimanda alla lettura del file README.md per il dettaglio esaustivo della semantica dei parametri supportati in linea di comando di entrambi i programmi.
Un esempio di uso del programma pmc3t_fit.py
Si supponga di avere a disposizione un dataset di training (ad esempio generato tramite pmc3t_gen.py come mostrato nel paragrafo precedente)
e si voglia che il MLP abbia tre layer nascosti rispettivamente con 200, 300 e 200 neuroni e che si voglia usare la funzione di attivazione sigmoid in uscita da tutti e tre i layer;
inoltre si vogliano eseguire 500 epoche di training con un batch size di 200 elementi usando l'algoritmo di ottimizzazione Adamax con un learning rate di 0.02
e la MeanSquaredError quale funzione di loss. Per mettere in azione tutto questo si esegua il seguente comando:
$ python pmc3t_fit.py \
--trainds mytrain.csv \
--modelout mymodel \
--hlayers 200 300 200 \
--hactivation sigmoid sigmoid sigmoid \
--epochs 500 \
--batch_size 200 \
--optimizer 'Adamax(learning_rate=0.02)' \
--loss 'MeanSquaredError()'mymodel conterrà il modello del MLP addestrato sul dataset mytrain.csv secondo i parametri passati in linea di comando.
Predizione
Come per l'addestramento anche questa funzionalità ha due implementazioni differenti secondo le due definizioni matematiche di curva parametrica: l'implementazione ufficiale è quella che effettua la predizione usando un solo MLP pre-addestrato che approssima la curva definita come funzione vettoriale.
Scopo della versione ufficiale è realizzata dal programma Python pmc3t_predict.py
è quello di applicare il modello di MLP generato tramite pmc3t_fit.py a un dataset in input (ad esempio il dataset di test generato tramite pmc3t_gen.py come mostrato in un paragrafo precedente);
l'esecuzione del programma produce in uscita un file csv con tre colonne (senza header): la prima colonna contiene i valori del parametro indipendente $t$ presi dal dataset in input
mentre la seconda, la terza e la quarta colonna contengono i valori predetti delle tre componenti, ovverosia i valori della predizione che approssimano la curva parametrica $f(t)$ corrispondenti al valore di $t$ della prima colonna.
Per ottenere l'usage della versione ufficiale del programma è sufficiente eseguire il seguente comando:
$ python pmc3t_predict.py --helpusage: pmc3t_predict.py [-h]
--model MODEL_PATH
--ds DATASET_FILENAME
--predictionout PREDICTION_DATA_FILENAMEUn esempio di uso del programma pmc3t_predict.py
Si supponga di avere a disposizione il dataset di test mytest.csv (ad esempio generato tramite pmc3t_gen.py come mostrato in un paragrafo precedente)
e il modello di MLP addestrato nella cartella mymodel (generato tramite pmc3t_fit.py come mostrato nell'esempio del paragrafo precedente); si esegua quindi il seguente comando:
$ python pmc3t_predict.py \
--model mymodel \
--ds mytest.csv \
--predictionout myprediction.csv
myprediction.csv conterrà l'approssimazione della curva parametrica iniziale.
Visualizzazione del risultato
Scopo del programma Python pmc3t_plot.py
è quello di visualizzare la curva della predizione sovrapposta alla curva del dataset iniziale (che sia quello di test o di training) e questo consente la comparazione visuale delle due curve.
Per ottenere l'usage del programma è sufficiente eseguire il seguente comando:
$ python pmc3t_plot.py --helpusage: pmc3t_plot.py [-h]
--ds DATASET_FILENAME
--prediction PREDICTION_DATA_FILENAME
[--savefig SAVE_FIGURE_FILENAME]Un esempio di uso del programma pmc3t_plot.py
Avendo a disposizione il dataset di test mytest.csv (ad esempio generato tramite pmc3t_gen.py come mostrato in un paragrafo precedente)
e il file csv della predizione (generato tramite pmc3t_predict.py come mostrato nel precedente paragrafo), per generare i due grafici si esegua il seguente comando:
$ python pmc3t_plot.py \
--ds mytest.csv \
--prediction myprediction.csvNota: Data la natura stocastica della fase di addestramento, i singoli specifici risultati possono variare. Si consideri di eseguire la fase di addestramento più volte.
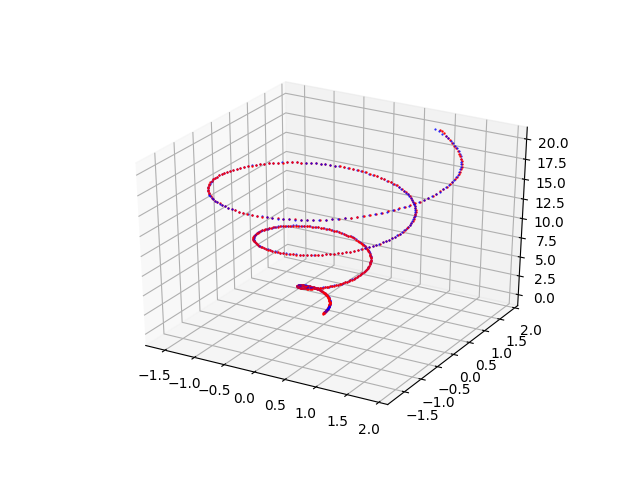
pmc3t_plot.py che mostra l'approssimazione effettuata dal MLP della spirale di Archimede nello spazio $f(t) = \begin{bmatrix} x(t)=\frac{1}{10} t \cos t \\ y(t)=\frac{1}{10} t \sin t \\ z(t)=t \\ \end{bmatrix}$Esempi di uso in cascata dei quattro programmi
Nella cartella parametric-curve-in-space-fitting/examples
ci sono cinque script shell che mostrano l'uso dei quattro programmi in cascata, con addestramento e predizione nella versione ufficiale, in varie combinazioni
di parametri (architettura del MLP, funzioni di attivazione, algoritmo, funzione di loss, parametri di prodedura di training).
Per mandare in esecuzione i cinque esempi nella versione ufficiale eseguire i seguenti comandi:
$ cd parametric-curve-in-space-fitting/examples
$ sh example1.sh
$ sh example2.sh
$ sh example3.sh
$ sh example4.sh
$ sh example5.sh
Download del codice completo
Il codice completo è disponibile su GitHub.
Questo materiale è distribuito su licenza MIT; sentiti libero di usare, condividere, "forkare" e adattare tale materiale come credi.
Sentiti anche libero di pubblicare pull-request e bug-report su questo repository di GitHub oppure di contattarmi sui miei canali social disponibili nell'angolo in alto a destra di questa pagina.