Approssimazione di funzioni tramite un Support Vector Regressor configurabile
Questo post tratta l'approssimazione di funzioni matematiche reali, sia scalari che vettoriali, a una o più variabili reali tramite il Support Vector Regressor senza scrivere codice ma agendo solo sulla linea di comando di script Python che implementano le funzionalità di:
- Configurazione del regressore e addestramento
- Predizione e calcolo dell'errore
Per quanto riguarda la generazione dei dataset sintetici di training e di test si farà uso dei seguenti strumenti (presenti nel repository):
-
fx_gen.pyper le funzioni generatrici scalari reali di una variabile reale $f(x) \colon [a,b] \to {\rm I\!R}$ -
fxy_gen.pyper le funzioni generatrici scalari reali di due variabili reali $f(x,y) \colon [a,b] \times [c,d] \to {\rm I\!R}$ -
pcm2t_gen.pyper le curve parametriche sul piano, quindi funzioni vettoriali reali $f(t) \colon [a,b] \to {\rm I\!R \times \rm I\!R}$ -
pmc3t_gen.pyper le curve parametriche nello spazio, quindi funzioni vettoriali reali $f(t) \colon [a,b] \to {\rm I\!R \times \rm I\!R \times \rm I\!R}$
-
fx_scatter.pyper le funzioni scalari reali di una variabile reale -
fxy_scatter.pyper le funzioni scalari reali di due variabili reali -
pmc2t_scatter.pyper le curve parametriche sul piano -
pmc3t_scatter.pyper le curve parametriche nello spazio
Configurazione del regressore e addestramento
In questo capitolo vengono presentati due programmi: fit_func_esvr.py e
fit_func_nusvr.py
che tecnicamente sono wrapper rispettivamente delle classi della libreria SciKit-Learn
sklearn.svm.SVR e
sklearn.svm.NuSVR
che implementano gli algoritmi epsilon-Support-Vector-Regressor e nu-Support-Vector-Regressor e
il fine dei due programmi è di consentire l'utilizzazione dei regressori sottostanti per approssimare funzioni
senza dover scrivere codice ma agendo solo sulla linea di comando.
Infatti tramite l'argomento --svrparams l'utente passa una serie di iper parametri per regolare il comportamento dell'algoritmo SVR sottostante
e altri per configurare l'apprendimento dello stesso.
Oltre ai parametri del regressore sottostante i due programmi supportano dei propri argomenti per consentire all'utente di passare
il dataset di training e il file ove salvare il modello addestrato.
Entrambi i programmi sono di tipo M.I.M.O., cioè Multiple Input Multiple Output:
sono progettati per approssimare una funzione della forma $f \colon \rm I\!R^n \to \rm I\!R^m$ utilizzando nell'implementazione
la classe sklearn.multioutput.MultiOutputRegressor.
Il formato dei dataset in ingresso è in formato csv (con header), con $n + m$ colonne, di cui le prime $n$ colonne contengono i valori delle $n$ variabili indipendenti e
le ultime $m$ contenenti i valori delle variabili dipendenti.
Usage del programma fit_func_esvr.py
Per ottenere l'usage del programma è sufficiente eseguire il seguente comando:
$ python fit_func_esvr.py --helpusage: fit_func_esvr.py [-h] [--version] --trainds TRAIN_DATASET_FILENAME
--outputdim NUM_OF_DEPENDENT_COLUMNS --modelout
MODEL_FILE [--dumpout DUMPOUT_PATH]
[--svrparams SVR_PARAMS]
fit_func_esvr.py fits a multiple-input multiple-output function dataset using
a configurable Epsilon-Support Vector Regressor
optional arguments:
-h, --help show this help message and exit
--version show program's version number and exit
--trainds TRAIN_DATASET_FILENAME
Train dataset file (csv format)
--outputdim NUM_OF_DEPENDENT_COLUMNS
Output dimension (alias the number of dependent
columns, that must be last columns)
--modelout MODEL_FILE
Output model file
--svrparams SVR_PARAMS
Parameters of Epsilon-Support Vector Regressor
constructor-
-h, --help: mostra l'usage del programma e termina l'esecuzione.
-
--version: mostra la versione del programma e termina l'esecuzione.
-
--trainds: percorso (relativo o assoluto) del file csv a $n+m$ colonne (con header) che contiene il dataset da utilizzare per il training;
questo file può essere generato in modo sintetico ad esempio tramite uno dei programmi di cui sopra
oppure essere un dataset del mondo reale che mappa $n$ variabili indipendenti su $m$ variabili dipendenti.
-
--outputdim:
il numero $n$ delle variabili indipendenti che devono corrispondere alle prime $n$ colonne del dataset csv;
il resto delle colonne a destra sono considerante di conseguenza la $m$ variabili dipendenti.
-
--modelout: percorso (relativo o assoluto) al file dove salvare il modello addestrato nel formato joblib (.jl).
-
--svrparams:
lista di parametri da passare all'algoritmo di regressione sottostante;
si veda la documentazione di
sklearn.svm.SVR.
Usage del programma fit_func_nusvr.py
Per ottenere l'usage del programma è sufficiente eseguire il seguente comando:
$ python fit_func_nusvr.py --helpusage: fit_func_nusvr.py [-h] [--version] --trainds TRAIN_DATASET_FILENAME
--outputdim NUM_OF_DEPENDENT_COLUMNS --modelout
MODEL_FILE [--dumpout DUMPOUT_PATH]
[--svrparams SVR_PARAMS]
fit_func_nusvr.py fits a multiple-input multiple-output function dataset using
a configurable Nu-Support Vector Regressor
optional arguments:
-h, --help show this help message and exit
--version show program's version number and exit
--trainds TRAIN_DATASET_FILENAME
Train dataset file (csv format)
--outputdim NUM_OF_DEPENDENT_COLUMNS
Output dimension (alias the number of dependent
columns, that must be last columns)
--modelout MODEL_FILE
Output model file
--svrparams SVR_PARAMS
Parameters of Nu-Support Vector Regressor constructor-
-h, --help: mostra l'usage del programma e termina l'esecuzione.
-
--version: mostra la versione del programma e termina l'esecuzione.
-
--trainds: percorso (relativo o assoluto) del file csv a $n$+$m$ colonne (con header) che contiene il dataset da utilizzare per il training;
questo file può essere generato in modo sintetico ad esempio tramite uno dei programmi di cui sopra
oppure essere un dataset del mondo reale che mappa $n$ variabili indipendenti su $m$ variabili dipendenti.
-
--outputdim:
il numero $n$ delle variabili indipendenti che devono corrispondere alle prime $n$ colonne del dataset csv;
il resto delle colonne a destra sono considerante di conseguenza la $m$ variabili dipendenti.
-
--modelout: percorso (relativo o assoluto) al file dove salvare il modello addestrato nel formato joblib (.jl).
-
--svrparams:
lista di parametri da passare all'algoritmo di regressione sottostante;
si veda la documentazione di
sklearn.svm.NuSVR.
Predizione e calcolo dell'errore
In questo capitolo viene invece presentato il programma predict_func.py
il cui scopo è quello di effettuare le predizioni su un dataset di test applicandolo a un modello e-SVR oppure nu-SVR addestrato precedentemente
rispettivamente tramite il programma fit_func_esvr.py o
fit_func_nusvr.py
sempre senza dover scrivere codice ma tramite la sola linea di comando.
Infatti tale programma supporta argomenti tramite i quali l'utente passa il modello addestrato precentemente, il dataset di test
e le misure di errore da calcolare tra la predizione e il valore vero.
Il formato dei dataset di test in ingresso è identico a quello dei programmi di training di cui sopra; ovviamente qui le ultime colonne
(quelle delle variabili dipendenti) vengono adoperate solo per confrontare i valori predetti con i valori veri calcolando le misure di errore passate.
Usage del programma predict_func.py
Per ottenere l'usage del programma è sufficiente eseguire il seguente comando:
$ python predict_func.py --helpusage: predict_func.py [-h] [--version] --model MODEL_FILE --ds DF_PREDICTION
--outputdim NUM_OF_DEPENDENT_COLUMNS --predictionout
PREDICTION_DATA_FILE
[--measures MEASURES [MEASURES ...]]
predict_func.py makes prediction of the values of a multiple-input multiple-
output function with a pretrained Standard Vector Regressor model
optional arguments:
-h, --help show this help message and exit
--version show program's version number and exit
--model MODEL_FILE model file
--ds DF_PREDICTION dataset file (csv format)
--outputdim NUM_OF_DEPENDENT_COLUMNS
Output dimension (alias the number of dependent
columns, that must be last columns)
--predictionout PREDICTION_DATA_FILE
prediction data file (csv format)
--measures MEASURES [MEASURES ...]
List of built-in sklearn regression metrics to compare
prediction with input dataset-
-h, --help: mostra l'usage del programma e termina l'esecuzione.
-
--version: mostra la versione del programma e termina l'esecuzione.
-
--model: percorso (relativo o assoluto) al file nel formato joblib (.jl) del modello generato da uno dei programmi di training sopra descritti.
-
--ds: percorso (relativo o assoluto) del file csv (con header) che contiene il dataset di input su cui calcolare la predizione.
-
--outputdim:
il numero $n$ delle variabili indipendenti che devono corrispondere alle prime $n$ colonne del dataset csv;
il resto delle colonne a destra sono considerante di conseguenza la $m$ variabili dipendenti.
-
--predictionout: percorso (relativo o assoluto) del file csv da generare che conterrà la predizione, ovverosia l'approssimazione della funzione applicata al dataset di input.
-
--measures: lista di misure da calcolare confrontando i valori veri del dataset di input e i valori predetti in uscita;
la lista delle metriche supportate è definita in SciKit Learn Regression Metrics.
Un esempio di uso di tutti i programmi
Si supponga di voler approssimare nell'intervallo $[-0.10,10.0]$ la seguente funzione $$f(x)=\frac {1}{2} x^3 - 2 x^2 - 3 x - 1$$ Tenendo presente che np è l'alias della libreria NumPy, questa si traduce in sintassi lambda body Python così:
0.5*x**3 - 2*x**2 - 3*x - 1$ python fx_gen.py \
--dsout mytrain.csv \
--funcx "0.5*x**3 - 2*x**2 - 3*x - 1" \
--xbegin -10.0 \
--xend 10.0 \
--xstep 0.01$ python fx_gen.py \
--dsout mytest.csv \
--funcx "0.5*x**3 - 2*x**2 - 3*x - 1" \
--xbegin -10.0 \
--xend 10.0 \
--xstep 0.0475A questo si intende effettuare una regressione tramite
fit_func_esvr.py
passando al regressore sottostante i seguenti argomenti: kernel: rbf, C: 100, gamma: 0.1, epsilon: 0.1;
quindi si esegua quindi il seguente comando:
$ python fit_func_esvr.py \
--trainds mytrain.csv \
--modelout mymodel.jl \
--outputdim 1 \
--svrparams "'kernel': 'rbf', 'C': 100, 'gamma': 0.1, 'epsilon': 0.1"Adesso si intende effettuare la predizione e il calcolo dell'errore usando le misure mean_absolute_error e mean_squared_error; quindi si esegua quindi il seguente comando:
$ python predict_func.py \
--model mymodel.jl \
--ds mytest.csv \
--outputdim 1 \
--predictionout mypred.csv \
--measures mean_absolute_error mean_squared_error--measures
e sono accettabili: la prima intorno a $0.4$ e la seconda intorno a $1.9$Nota: Data la natura stocastica della fase di addestramento, i singoli specifici risultati possono variare. Si consideri di eseguire la fase di addestramento più volte.
Infine si intende effettuare la visualizzazione comparata del dataset di test con la predizione; si esegua perciò il seguente comando:
$ python fx_scatter.py \
--ds mytest.csv \
--prediction mypred.csv \
--title "e-svr ('kernel': 'rbf', 'C': 100, 'gamma': 0.1, 'epsilon': 0.1)" \
--xlabel "x" \
--ylabel "y=1/2 x^3 - 2x^2 - 3x - 1"Nota: Data la natura stocastica della fase di addestramento, i singoli specifici risultati possono variare. Si consideri di eseguire la fase di addestramento più volte.
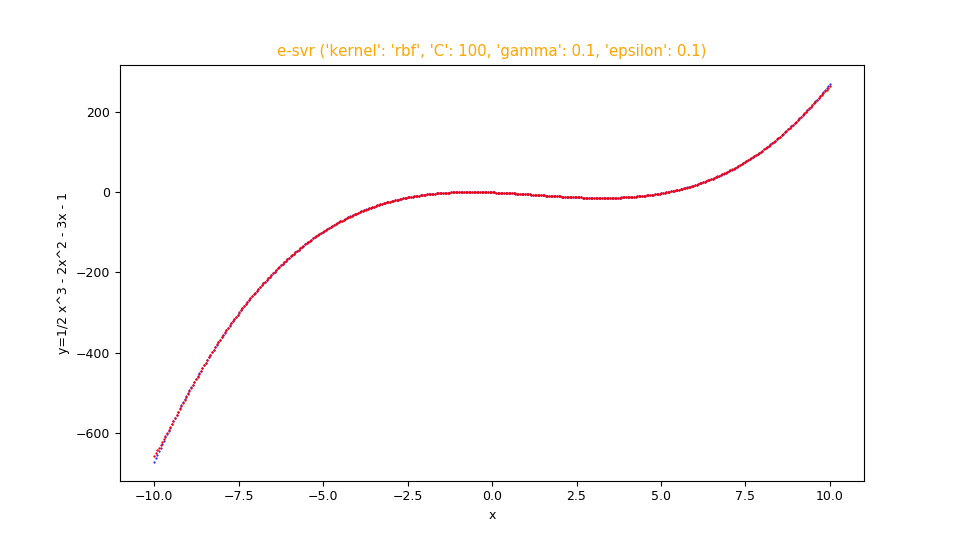
fx_scatter.py che mostra l'approssimazione in sovraimpressione in rosso della funzione $f(x)=\frac {1}{2} x^3 - 2 x^2 - 3 x - 1$
e la funzione originale sottostante in blu.
Se si intende invece effettuare una regressione tramite fit_func_nusvr.py
passando al regressore sottostante i seguenti argomenti: C: 10, gamma: auto
si esegua quindi il seguente comando:
$ python fit_func_nusvr.py \
--trainds mytrain.csv \
--modelout mymodel.jl \
--outputdim 1 \
--svrparams "'C': 10, 'gamma': 'auto'"predict_func.py con gli stessi parametri di sopra
e infine per generare i grafici a dispersione eseguire il seguente comando:
$ python fx_scatter.py \
--ds mytest.csv \
--prediction mypred.csv \
--title "nu-svr ('C': 10, 'gamma': 'auto')" \
--xlabel "x" \
--ylabel "y=1/2 x^3 - 2x^2 - 3x - 1"-
one-variable-function/svr/examplesper esempi sull'approssimazione di funzioni scalari reali di una variabile reale tramite SVR. -
two-variables-function/svr/examplesp per esempi sull'approssimazione di funzioni scalari reali di due variabili reali tramite SVR. -
parametric-curve-on-plane/svr/examplesper esempi sull'approssimazione di curve parametriche sul piano tramite SVR. -
parametric-curve-in-space/svr/examplesper esempi sull'approssimazione di curve parametriche nello spazio tramite SVR.
Download del codice completo
Il codice completo è disponibile su GitHub.
Questo materiale è distribuito su licenza MIT; sentiti libero di usare, condividere, "forkare" e adattare tale materiale come credi.
Sentiti anche libero di pubblicare pull-request e bug-report su questo repository di GitHub oppure di contattarmi sui miei canali social disponibili nell'angolo in alto a destra di questa pagina.